LA STORIA DELLA GENOMICA: COME NASCE, COME SI SVILUPPA E COSA CI RISERVA IN FUTURO
di Sergio Barocci – Università di Genova per la terza età e divulgatore scientifico.
Incremento delle conoscenze sugli acidi nucleici (parte II)
Il dogma centrale della biologia molecolare

Nel 1958, venne proposta da Francis Crick in una importante comunicazione la famosa “teoria della tripletta” nota come il “dogma centrale della biologia molecolare” che fissava le relazioni tra DNA, RNA e proteine:
a) il DNA è il portatore dell’informazione genetica; b) il processo di replicazione crea nuove coppie di DNA; c) il processo della trascrizione crea RNA utilizzando le informazioni del DNA; d) il processo di traduzione crea le proteine usando le informazioni dell’RNA.
Nel frattempo, sempre nel 1958, Francois Jacob (1920–2013) e Jacques Monod (1910–1976) intuirono l’esistenza di un intermediario instabile, l’RNA messaggero, la molecola che funge da collegamento tra l’informazione contenuta nel DNA e quella espressa nelle proteine cioè la molecola che portava l’informazione al DNA dal nucleo della cellula alla fattoria di proteine (i ribosomi) nel citoplasma.
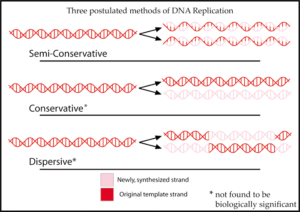
Il meccanismo semiconservativo della replicazione del DNA

Nel 1958 ci fu anche la conferma finale del meccanismo di replicazione basato sulla struttura a doppia elica fornita dall’esperimento di Matthew Meselson (Denver, 24maggio 1930 – ) e Franklin Stahl (Boston, 8ottobre 1928 – ) – esperimento fondamentale per determinare il meccanismo semiconservativo di replicazione del DNA – definito anche come il più bel esperimento della biologia.
Prima di Watson, Crick e Wilkins, il Premio Nobel per la Medicina fu assegnato nel 1959 al biochimico statunitense Arthur Kornberg (1918–2007), per aver scoperto all’Università di Stanford nel 1957 un enzima cellulare, chiamato DNA polimerasi, che lega fra loro le due eliche e che in natura svolge diverse funzioni comprese la replicazione e la riparazione del DNA. Questo enzima può allungare un corto oligonucleotide iniziatore o “primer” aggiungendo un nucleotide per volta alla sua estremità 3’ (denominata così semplicemente per convenzione chimica) ma solo se il primer è ibridizzato o legato al filamento complementare detto filamento stampo o “template”.
Kornberg e la scoperta della DNA polimerasi
Il nucleotide attaccato è complementare alla base che si trova nella posizione corrispondente sul filamento stampo.
Ad esempio se quest’ultima è una A, la polimerasi attacca una T; se la base sul filamento è una G l’enzima attacca una C. Ripetendo questo processo, la polimerasi può estendere l’estremità 3’ del primer sino al termine 5’ dello stampo.
In una doppia elica di DNA ciascun filamento serve da stampo all’altro durante la replicazione e la riparazione. La correzione degli errori avviene ad opera dell’attività 3’ esonucleasica della DNA polimerasi grazie alla quale l’enzima è in grado di riconoscere l’incorporazione di un nucleotide sbagliato, di eliminarlo e di formare un corretto appaiamento con il corrispondente nucleotide presente sullo stampo.
doppia elica e codice genetico

Una volta compresi i dettagli della struttura della doppia elica, rimaneva da decifrare il codice genetico: come venivano specificati, usando un alfabeto di sole quattro lettere, i venti amminoacidi di cui sono costituite tutte le proteine?
Questa ricerca, in definitiva, divenne molto fruttuosa in quanto furono fatte tre scelte ispirate:
1) quali erano i precursori attivati del DNA? Correttamente, Kornberg dedusse che i deossiribonucleotidi 5’ trifosfato erano gli intermedi attivati;
2) quale era il criterio di sintesi del DNA? Si riteneva che la quantità di DNA sintetizzato doveva essere piccola e che pertanto era essenziale un metodo di dosaggio molto sensibile per cui si utilizzarono come precursori dei nucleotidi radioattivi. L’incorporazione di questi precursori nel DNA era rivelata misurando la radioattività di un precipitato acido della miscela di incubazione. (Il DNA viene precipitato dall’acido tricloacetico mentre i nucleotidi rimangono in soluzione);
L’esprimento di Kornberg

3) quale specie di cellule si dovevano analizzare? Si utilizzarono delle cellule batteriche dopo esperimenti iniziali di cellule di animali. Il batterio Escheria coli fu scelto in quanto possedeva un ampio tempo di replicazione di soli 20’ e poteva essere ottenuto in grande quantità e come ci si aspettò questo batterio rappresentava un’ottima fonte di enzimi che sintetizzavano il DNA.
L’esperimento di Kornberg consisteva in un estratto di E. coli che conteneva come enzima la DNA polimerasi che aggiunse ad una soluzione salina contenente molecole di DNA e quattro deossinucleotidi comprendenti timina marcata radioattivamente (marcata con 14C).
Dopo incubazione a temperatura ambiente, la deossitimidina comparve in lunghi polinucleotidi, segnalando che la DNA polimerasi aveva funzionato generando nuovo DNA.
DNA POLIMERASI e codoni

Ora questo enzima è chiamato DNA polimerasi I perché da allora sono state isolate diverse DNA polimerasi. La DNA Polimerasi I non è l’enzima principale della replicazione, il cui ruolo spetta alla DNA polimerasi III (nota per questo anche come replicasi).
Essa svolge invece le seguenti funzioni: a) funzione di riparazione dei danni del DNA, riempendo brevi regioni a singolo filamento; b) ruolo accessorio durante la replicazione, consistente nel sostituire i ribonucleotidi (RNA) con deossiribonucleotidi (DNA), sul filamento lagging ( filamento ritardato: copia di DNA sintetizzata sullo stampo che ha direzione 5’→3’).
Nel 1961 il biologo sudafricano Sydney Brenner (1927 – 2019) e Francis Crick scoprirono che inserire o eliminare una o due lettere nel DNA produceva un effetto devastante ma inserirne o eliminarne tre no, e cosi capirono che nel primo caso si riscrivevano tutte le parole, mentre nel secondo se ne perdeva solo una: le parole del codice genetico, dette «codoni», erano dunque di tre lettere.
CODICE GENETICO E TRIPLETTE

E poiché con un alfabeto di quattro lettere si possono fare 64 codoni distinti, il codice doveva essere ridondante.
La dimostrazione che il codice genetico fosse basato su triplette di basi non sovrapposte, permise al biochimico indiano Har Gobind Khorana (9gennaio 1922 – 9novembre 2011), ai biochimici statunitensi Robert William Holley (28gennaio 1922 – 11febbraio 1993) e Marshall Warren Nirenberg (10aprile 1927 – 15gennaio 2010) di decifrarlo.
Nirenberg scoprì, infatti, che uno dei più semplici segmenti di DNA, costituito di sole A (adenina), produceva un particolare amminoacido (la fenilalanina): il codone corrispondente (o “parola” fatta da nucleotidi), dunque, doveva essere la tripletta di basi AAA, e insieme a Khorana, che mise a punto tecniche chimiche per fabbricare segmenti di DNA consistenti di un solo codone e a Holley per aver individuato la struttura del tRNA responsabile del trasporto dell’alanina ricevettero il Premio Nobel per la Medicina nel 1968 per la loro interpretazione del codice genetico e della sua funzione nella sintesi proteica.
LA BASE DELLA BIOLOGOGIA MOLECOLARE

Queste scoperte sono state la base della moderna biologia molecolare. Il passaggio dal DNA alle proteine non era però diretto ma mediato da una seconda forma di acido nucleico, chiamata RNA. Quindi è tra il periodo che va dal 1961 al 1966 che venne decifrato il codice genetico, cioè venne stabilito il rapporto tra le 64 triplette possibili a partire dalle 4 basi nucleotidiche del DNA e i 20 aminoacidi che formavano le proteine.
Infatti, c’è da considerare che già Crick, nel 1958 nel proporre il dogma centrale della biologia molecolare, in cui l’informazione genetica andava a senso unico, dal DNA all’RNA alle proteine, spiegò questo strano meccanismo, adducendo che l’uovo (il DNA) veniva necessariamente prima della gallina (le proteine), ipotizzando quindi che l’RNA si stata la prima molecola genetica, in un’epoca in cui la vita era basata solo su di essa mentre il DNA si sarebbe sviluppato successivamente, probabilmente in risposta all’instabilità dell’RNA.
DNA, RNA E PROTEINE

Però, per scoprire il modo in cui le proteine venivano sintetizzate, si dovette aspettare lo sviluppo di estratti crudi (cell-free), in grado di eseguire tutti i passaggi necessari per la sintesi.
Grazie a questo metodo Paul Charles Zamecnik (1912–2009) e collaboratori scoprirono che gli amminoacidi vengono uniti alle molecole che oggi chiamiamo RNA transfer (tRNA) da una classe di enzimi denominati amminoacilsintetasi, prima della loro incorporazione nelle proteine.
L’RNA transfer rappresenta circa il 10% di tutto l’RNA cellulare. Oggi si conosce che ogni tRNA si contiene una sequenza di basi adiacenti (l’anticodone) che si lega in maniera specifica a gruppi di basi contigue (codoni) lungo lo stampo di RNA durante la sintesi proteica.
VARI TIPI DI RNA

In ogni caso e, circa l’85% dell’RNA cellulare si trova nei ribosomi e dato che il loro numero aumenta notevolmente nelle cellule che hanno un’intensa attività di sintesi proteica, si pensò inizialmente che l’RNA ribosomiale (rRNA) fosse lo stampo per ordinare gli amminoacidi. Tuttavia, grazie all’utilizzo di cellule infettate con il fago T4, si scoprì che lo stampo per ordinare gli amminoacidi è l’RNA messaggero (mRNA), dato che trasporta l’informazione del DNA ai siti ribosomiali per la sintesi proteica.
Mentre si scopriva l’RNA messaggero, i biochimici statunitensi Jerard Hurwitz (1928 – 2019) e Samuel B. Weiss isolarono in maniera indipendente il primo degli enzimi in grado di trascrivere l’RNA a partire da uno stampo di DNA. Questi enzimi, chiamati RNA polimerasi, funzionano solo in presenza di DNA, che funge da stampo su cui si formano le catene a singolo filamento di RNA.
UOVO E GALLINA

Nei batteri, lo stesso enzima produce ciascuna delle classi maggiori di RNA (ribosomiale, transfer e messaggero), usando appropriati segmenti di DNA cromosomico stampo. Nel 1983 i biochimici statunitensi Tomas Robert Cech (1947 – ) e Sidney Altman (7maggio 1939 – 5aprile 2022) diedero la prima conferma che l’RNA era una sorta di «uovo-gallina» autocatalizzante, e ottennero il premio Nobel per la chimica nel 1989.
Una volta compreso l’alfabeto e le parole del codice genetico, rimaneva da leggere l’intero libro: il genoma delle varie specie, uomo compreso. I capitoli del libro presero il nome di geni, e la scoperta di come si attivavano e si disattivavano in un batterio intestinale (E. coli) valsero il premio Nobel del 1965 a Monod e a Jacob: una coppia la cui popolarità rivaleggiava con quella di Watson e Crick, grazie anche ai loro rispettivi libri “Il caso e la necessità” (J. Monod) e “La logica del vivente“ ed. Einaudi, 1971 (F. Jacob ).
Il significato di codice genetico degenerato
Il codice genetico viene definito degenerato in quanto più codoni possono codificare per uno stesso aminoacido. Le triplette che codificano lo stesso aminoacido presentano di solito le prime due posizioni conservate, mentre la terza varia.
clicca per andare alla terza parte
Bibliografia:
- Holley R.W. et al. (1965) . “Nucleotide Sequences In The Yeast Alanine Transfer Ribonucleic Acid” . J Biol Chem. 240 (5): 2122–8.
- Smith H & Wilcox KW (1970). “A Restriction enzyme from Hemophilus influenza : I. Purification and general properties“. Journal of Molecular Biology. 51 (2): 379 – 391.
- Josse J, Kaiser A.D., Konberg A. (1961)” Enzymatic synthesis of deoxyribonucleic acid. VIII. Frequencies of nearest neighbor base sequences in deoxyribonucleic acid” J Biol Chem 236 : 864 – 875.
- Khorana H.G. ( 1954) “Carbodiimides. Part V. A novel synthesis of adenosine di – and triphosphate and P1, P2 diadenosine – 5’ – pyrophosphate “ J Am Chem Soc 76: 3517 – 3522.
- Khorana H.G. (1960) “Synthesis of nucleotides , nucleotide co-enzymes and polynucleotides “ Fed Proceedings 19: 931 – 94.
- Nirenberg M.& Matthaei J.M. (1961) “The dependence of cell- free protein synthesis in E. Coli upon naturally occurring or synthetic polyribonucleotides” PNAS 47(10): 1588 – 1602
- Nirenberg M.& Leder P. (1964) “RNA Codewords and Protein Synthesis : the effect of trinucleotides upon the binding of sRNA to ribosomes” Science 145(3639): 1399 – 1407.