Storia della reazione a catena della polimerasi (PCR)
- Articolo del prof. Sergio Barocci – Università di Genova per la terza età.
sviluppo della reazione a catena della polimerasi
 |

L’articolo di J. Watson e F. Crick: “Struttura molecolare degli acidi nucleici“, Nature 25 aprile 1953. |
Lo sviluppo della reazione a catena della polimerasi (PCR) è stato un importante passo avanti nel mondo scientifico. Nel corso degli anni, la tecnica si è molto evoluta oltre i confini del suo semplice design iniziale aprendo importanti strade per i ricercatori.
Nel giro di oltre 40 anni dalla sua scoperta, la PCR è diventata la base per diversi protocolli di biologia molecolare costituendo, inoltre, la base del Progetto Genoma Umano.
La sua storia, come tutti i principali sviluppi tecnologici della scienza, è stata segnata da controversie, rivendicazioni e contro – affermazioni, alcune delle quali sono risultate, ancora, irrisolte. Questa rassegna cerca di raccontare quegli sviluppi chiave che hanno condotto alla sua invenzione.
Sviluppi nella struttura del DNA
Il 25 Aprile del 1953, James Watson e Francis Crick pubblicavano su Nature un modello strutturale del DNA che presentava due filamenti di DNA che correvano in direzioni opposte come una doppia elica.
Neanche un decennio dopo, esattamente nel 1962, essi, insieme a Maurice Wilkins, responsabile del laboratorio del King’s College London in cui avvengono le scoperte, vengono insigniti del Premio Nobel per la Medicina. Tale riconoscimento non fu ,invece, mai ricevuto dalla biochimica Rosalind Franklin pioniera della cristallografia a raggi X che morì prematuramente nel 1958, all’età di 38 anni, a causa di un tumore ovarico probabilmente provocato dal massiccio utilizzo di raggi X.
Riparazione del DNA e attività ripetuta della polimerasi

A partire dalla metà degli anni ’50, il biochimico statunitense Arthur Konberg iniziava a studiare il meccanismo della replicazione del DNA e nel 1957 identificava la prima DNA polimerasi. Questo enzima era però limitato perché creava DNA in una sola direzione e per dare inizio alla sua replicazione aveva bisogno di un primer (un primer è una breve sequenza di acidi nucleici, utilizzata per dare inizio alla replicazione del DNA). Per questa scoperta, nel 1959, veniva insignito del Premio Nobel per la Medicina e Fisiologia.
Nel 1971, Gobind Khorana, biochimico indiano naturalizzato statunitense, vincitore del premio Nobel nel 1968 per il suo ruolo nella scoperta del codice genetico, avviava la strada, con il suo team di ricercatori, ad un grande progetto lavorando sulla sintesi della riparazione del DNA.
Cercando di copiare segmenti genici

Per raggiungere questo obiettivo, Khorana cercò di semplificare la sintesi genica creando e utilizzando oligonucleotidi di DNA sintetico. Questi oligonucleotidi sequenza-specifici utilizzati sia come elementi costitutivi del gene, sia come primer e modelli artificiali, dovevano aiutare la DNA polimerasi a copiare i segmenti genici desiderati. Tuttavia, sebbene questo processo fosse simile alla PCR nell’utilizzo di applicazioni ripetute di DNA polimerasi, esso impiegava solo un singolo complesso primer-stampo che, però, non portava all’amplificazione esponenziale osservata nella PCR.
Nello stesso periodo, sempre intorno al 1971, il biochimico norvegese Kjell Kleppe, un ricercatore del laboratorio di Khorana alla University of Wisconsin-Madison, utilizzava un sistema a due primer, simile alla PCR, che doveva condurre alla replicazione del segmento specifico di DNA desiderato.
Il sistema di Kleppe

La speranza era quella di ottenere due strutture, ciascuna delle quali contenente l’intera lunghezza del filamento del modello complessato con il primer.
Per completare il processo di replicazione della riparazione veniva, poi, aggiunta la DNA polimerasi. Il risultato ottenuto era quello di due molecole del duplex originale. L’intero ciclo, poteva essere ripetuto, aggiungendo ogni volta, una nuova quantità di enzima.
Sempre nel 1971 veniva fondata a Berkeley in California, un’azienda biotecnologica, la Cetus Corporation che inizialmente si occupava di progetti che coinvolgevano principalmente la clonazione e l’espressione di geni umani, oltre allo sviluppo di test diagnostici per le mutazioni genetiche. In seguito, questa azienda diventerà la sede dove si effettueranno gran parte delle ricerche che condurranno alla creazione della PCR.
La ricerca presso la Cetus Corporation

Alla Cetus, nel 1979, il biochimico Kary Mullis incominciò a lavorare sulla sintesi di oligonucleotidi che dovevano essere utilizzati per vari progetti di ricerca come la produzione di sonde per lo screening di geni clonati, di primer per il sequenziamento del DNA e per la sintesi di cDNA (DNA complementare) e di elementi costitutivi per varie tecniche di biologia molecolare. Mullis, originariamente sintetizzò questi oligonucleotidi manualmente ma poi incominciò ad interessarsi anche ad alcuni prototipi di sintetizzatori automatici.
Nel 1977 il chimico britannico Frederick Sanger identificava, nel frattempo, una metodologia per determinare la sequenza del DNA, che impiegava un oligonucleotide primer, una DNA polimerasi e dei precursori nucleotidici modificati che bloccavano l’ulteriore estensione del primer in maniera dipendente dalla sequenza. Grazie a questa tecnologia di sequenziamento, Sanger veniva insignito del Premio Nobel per la Chimica nel 1980.
Analisi dei prodotti PCR

Nei suoi primi esperimenti, Mullis non prevedeva cicli termici in quanto sperava che la DNA polimerasi continuasse a replicarsi da sola. Ma, nel 1984, conducendo esperimenti successivi con ripetuti cicli termici, egli notò che essi mostravano la capacità di amplificare il DNA genomico.
Il prodotto di amplificazione cioè l’aumento della quantità dei segmenti di DNA desiderati venne confermato dalla metodologia del Southern Blotting. La tecnica del Southern blotting – intitolata al suo inventore, il biologo britannico Edwin Mellor Southern, che l’ha pubblicata nel 1975 – è usata in laboratorio per rivelare la presenza di specifiche sequenze di DNA in una miscela complessa.
Taq DNA polimerasi
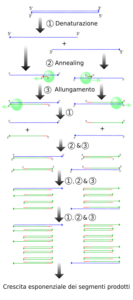
Nel frattempo, la PCR fu utilizzata oltre che per la clonazione e per il sequenziamento del DNA amplificato anche per altre applicazioni. Si incominciarono così a progettare nuovi primer e sonde, che rendevano la reazione più specifica fino a quando i risultati diventarono più evidenti all’elettroforesi su gel di agarosio.
Un altro importante passo avanti nella DNA polimerasi avvenne nel 1969, quando il microbiologo statunitense Thomas D. Brock riferì l’isolamento di Thermus aquaticus, una nuova specie di batterio termofilo trovata nelle sorgenti termali del Parco Nazionale di Yellowstone. La DNA polimerasi di questo batterio, chiamata Taq Polimerasi, era in grado di resistere a temperature molto elevate superiori a 75°, a differenza di altre polimerasi rese disponibili in quel momento.
Dopo un paio di tentativi falliti per il suo isolamento, due ricercatori della Cetus, Susanne Stoffel e David Gelfand, finalmente riuscirono nell’autunno del 1985 ad isolare la Taq polimerasi e, poco tempo dopo, Randy Saiki dimostrò che la Taq polimerasi era l’enzima ideale per il processo della PCR. Con i brevetti presentati, il lavoro proseguì per segnalare la PCR alla comunità scientifica generale.
Perkin-Elmer e Cetus sviluppano macchine PCR

Mullis e il suo gruppo pubblicarono tra il 1985 e il 1986 alcuni articoli sulla PCR e sulle sue applicazioni nelle principali riviste scientifiche e, nell’ottobre del 1990, rilasciato un brevetto per la PCR che utilizzava la Taq polimerasi.
Alla fine del 1985, l’azienda statunitense Perkin-Elmer e la Cetus formarono una joint venture per sviluppare reagenti e strumenti per la tecnica PCR. Seguì la produzione di complessi termociclatori cioè macchine PCR costruite per eseguire le amplificazioni basate su Klenow (frammento proteico prodotto quando la DNA polimerasi I di E. Coli subisce un clivaggio enzimatico e che possiede una attività polimerasica 5’→3’ e una attività esonucleasica 3′ → 5 ma non esonucleasica 5’→3’ per la rimozione dei nucleotidi già incorporati e per analisi di controllo) ma mai commercializzati, mentre la produzione di macchine PCR basate su Taq e la “DNA polimerasi AmpliTaq” si rese disponibile commercialmente nel novembre 1987.
Il Premio Nobel per Kary Mullis

Il 13 ottobre del 1993, Mullis, che nel frattempo aveva lasciato la Cetus nel 1986, è stato insignito del Premio Nobel per la Chimica.
Nel dicembre 1989, la Taq Polimerasi è stata nominata “Molecola dell’anno” dalla rivista Science. L’articolo sulla Taq PCR è diventato, in seguito, la pubblicazione più citata in biologia e la PCR ha rappresentato oltre il 3% di tutte le citazioni su PubMed.
La PCR è stata utilizzata per quantificare l’HIV nel sangue nella primavera del 1985. Verso la metà del 1987 era disponibile un test valido e la PCR è stata utilizzata per studiare l’impatto dei farmaci antivirali e anche per lo screening dei campioni di sangue dei donatori per l’HIV.
Le applicazioni della PCR

Nell’ottobre 1985, invece, la PCR è utilizzata per analizzare l’anemia falciforme, nella sua prima applicazione clinica e nel 1986 per l’analisi delle prove criminali. Tuttavia, solo nel 1989 sono stati ideati test di DNA Fingerprinting altamente sensibili basati sulla tecnica PCR, rendendola parte integrante delle indagini penali.
Nel 1987, il DNA di una ciocca di capelli umani è stato amplificato utilizzando la PCR e questo ha confermato la capacità della PCR di amplificare il DNA presente in campioni degradati parte delle prove forensi, mentre la Mutiplex-PCR è stata descritta per analizzare i prodotti di ricombinazione meiotica nel 1989. Queste amplificazioni a singola copia si sono poi rivelate cruciali per lo studio del DNA e della genotipizzazione.
Conclusioni
In breve, la tecnica della PCR, come oggi la conosciamo, è stata concettualizzata e sviluppata negli anni ’80 dal biochimico statunitense Kary Mullis e dai suoi colleghi della Cetus Corporation. L’isolamento e la purificazione delle Taq polimerasi termostabili hanno portato all’automazione di questa tecnologia, inizialmente lenta e laboriosa, e allo sviluppo di termociclatori PCR programmabili che l’hanno resa ampiamente utilizzata a molti livelli di biologia e chimica.
Sebbene K. Mullis abbia inventato la PCR, le sue applicazioni di successo in diversi campi sono state anche il risultato di un duro lavoro da parte di altri ricercatori Cetus. Inoltre, ci sono state anche delle sfide ai brevetti PCR detenuti da Mullis sulla base dei primi lavori di ricerca eseguiti da Khorana e dal suo team negli anni ’60 e ’70. Tuttavia, grazie alla sua straordinaria versatilità, oggi la tecnica della PCR viene utilizzata in diversi campi come le scienze forensi, gli studi ambientali, la tecnologia alimentare e la medicina diagnostica.
L’enorme progresso nel corso degli anni nella nostra comprensione del genoma degli esseri umani e di molte altre specie non sarebbe stato possibile senza la straordinaria ma semplice tecnica chiamata PCR.
Bibliografia:
- Mullis K.F. , Faloona F. , Scharf S. et al. (1986 ) “Specific enzymatic amplification of DNA in vitro : The polymerase chain reaction“ . Cold Spring Harbor Symposia on Quantitative Biology, vol. 51, pp 263 –273
- John M. Bartlett J.M. & Stirling D. “A Short History of the Polymerase Chain Reaction” Springer Nature Experiments 2003
- Pai – Dhungat J (2019) “Kary Mullis-inventor of PCR” J Assoc Physicians India 67(9) : 96.
- https://it.wikipedia.org/wiki/Reazione_a_catena_della_polimerasi
- https://it.wikipedia.org/wiki/Kary_Mullis